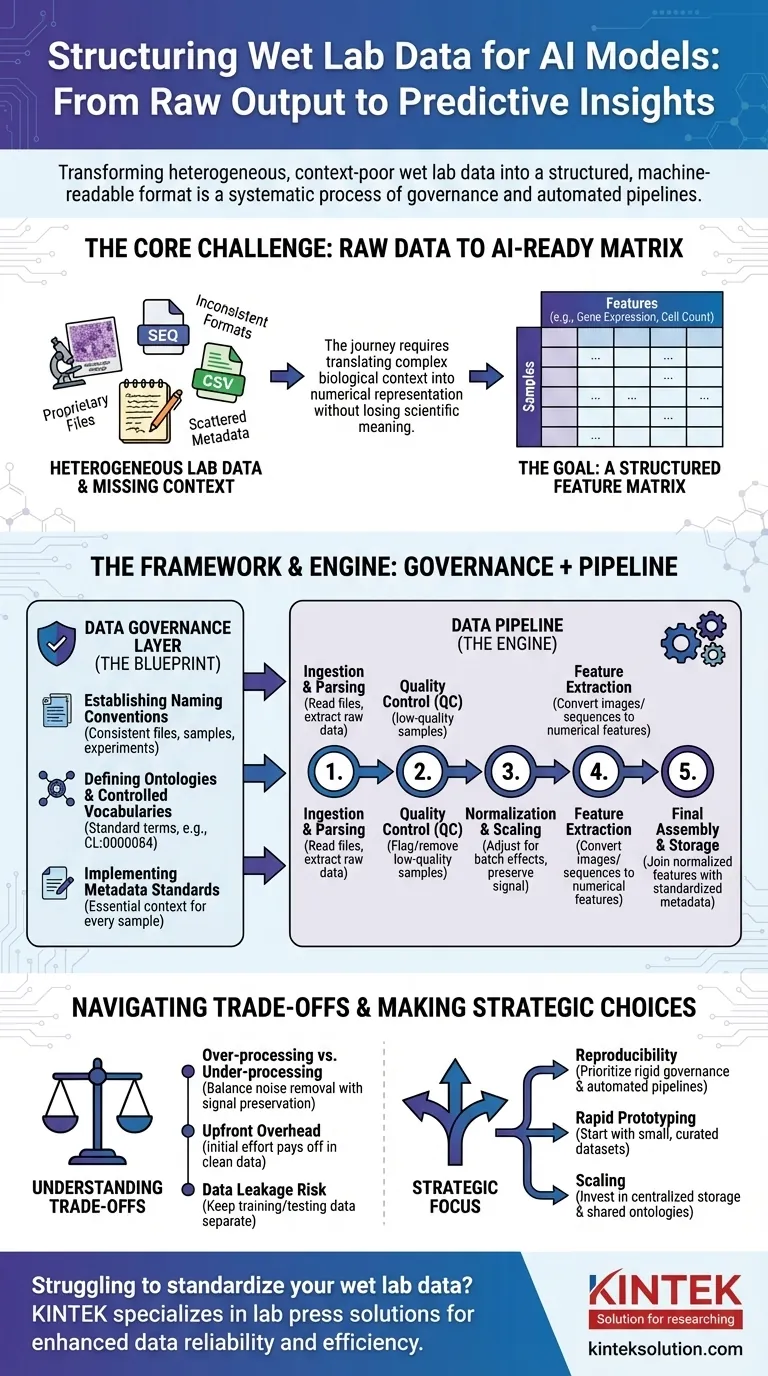
Um Nasslabor-Daten für KI vorzubereiten, müssen Sie diese aus ihrem rohen, oft inkonsistenten Zustand in ein strukturiertes, maschinenlesbares Format umwandeln. Dies ist kein einzelner Schritt, sondern ein systematischer Prozess, der Data Governance zur Schaffung klarer Regeln und anschließend Datenpipelines umfasst, die die Bereinigung, Normalisierung und Strukturierung roher experimenteller Ergebnisse in ein konsistentes Format automatisieren, das für das Modelltraining geeignet ist.
Die Kernherausforderung besteht nicht einfach darin, Dateien neu zu formatieren. Es geht darum, den komplexen biologischen Kontext – wie experimentelle Bedingungen, Probenhistorie und Messtechniken – systematisch in eine strukturierte, numerische Darstellung zu übersetzen, von der ein KI-Modell lernen kann, ohne dabei wichtige wissenschaftliche Bedeutungen zu verlieren.

Das Kernproblem: Von Rohdaten zu KI-tauglichen Daten
Der Weg vom Labortisch zu einem prädiktiven Modell ist voller Datenherausforderungen. Die Rohdaten wissenschaftlicher Instrumente sind selten oder nie direkt für die Verwendung in einem KI-Algorithmus bereit.
Die Heterogenität von Labordaten
Nasslabor-Daten liegen in einer Vielzahl von Formaten vor. Dies reicht von proprietären Dateien von Sequenziergeräten und Mikroskopen bis hin zu einfachen CSV-Dateien von Plattenlesegeräten, die jeweils ihre eigene Struktur und Eigenheiten haben.
Ein KI-Modell erfordert jedoch ein einheitliches Format.
Der Fluch des fehlenden Kontexts
Kritische Informationen, oder Metadaten, sind oft verstreut. Sie könnten in einem Laborbuch eines Wissenschaftlers, einer separaten Tabelle oder einfach in dessen Kopf enthalten sein. Ohne diesen Kontext (z. B. welches Medikament angewendet wurde, die Temperatur, die verwendete Zelllinie) sind die numerischen Daten bedeutungslos.
Das Ziel: Eine Merkmalsmatrix
Letztendlich benötigen die meisten KI-Modelle Daten in Form einer Merkmalsmatrix. Dies ist eine einfache Tabelle, bei der die Zeilen einzelne Proben (z. B. ein Patient, ein Zellkultur-Well) und die Spalten Merkmale (z. B. Genexpressionsniveaus, Zellmorphologiemessungen, Proteinkonzentrationen) darstellen.
Ein Rahmenwerk für die Standardisierung: Die Data-Governance-Schicht
Bevor Sie automatisierte Pipelines erstellen können, müssen Sie Regeln festlegen. Dies ist Data Governance – der Bauplan, der Konsistenz über alle Experimente und Teams hinweg gewährleistet. Es ist der wichtigste und oft übersehene Schritt.
Festlegen von Namenskonventionen
Eine einfache, aber wirkungsvolle Regel ist die Durchsetzung eines konsistenten Benennungsschemas für Dateien, Proben und Experimente. Dadurch können Daten programmatisch von ihrem Ursprung bis zur endgültigen Analyse verknüpft und nachverfolgt werden.
Definieren von Ontologien und kontrollierten Vokabularen
Eine Ontologie bietet einen Standardsatz von Begriffen zur Beschreibung biologischer Einheiten. Anstatt beispielsweise „T-Zelle“, „T-Lymphozyt“ und „Tcell“ zuzulassen, erzwingt ein kontrolliertes Vokabular einen einzigen Begriff, wie z. B. CL:0000084 aus der Zell-Ontologie.
Dies verhindert Mehrdeutigkeiten und stellt sicher, dass Daten aus verschiedenen Experimenten wirklich vergleichbar sind.
Implementierung von Metadatenstandards
Sie müssen die Mindestmetadaten definieren, die für jede einzelne Probe erfasst werden müssen. Dazu gehören häufig Probenquelle, experimentelle Bedingungen, Instrumenteneinstellungen und Datum. Diese Regel stellt sicher, dass kein Datenpunkt zu einer verwaisten, von ihrem Kontext getrennten Einheit wird.
Der Motor der Transformation: Aufbau der Datenpipeline
Sobald die Governance-Regeln festgelegt sind, können Sie eine Datenpipeline erstellen. Dies ist eine Reihe automatisierter Softwareschritte, die Rohdaten in die endgültige, KI-taugliche Merkmalsmatrix umwandeln.
Schritt 1: Datenerfassung und Parsing
Die erste Aufgabe der Pipeline besteht darin, die Rohdatendateien zu finden und zu lesen. Dieser Schritt umfasst das Schreiben spezifischer Parser für das Ausgabeformat jedes Instruments, um die primären Messungen und alle zugehörigen Metadaten zu extrahieren.
Schritt 2: Qualitätskontrolle (QC)
Nicht alle Daten sind gute Daten. Die Pipeline sollte automatisch Proben von geringer Qualität anhand vordefinierter Metriken kennzeichnen oder entfernen, wie z. B. niedrige Zellzahlen bei einem Bildgebungsexperiment oder schlechte Auslesequalität von einem Sequenziergerät.
Schritt 3: Normalisierung und Skalierung
Messungen von verschiedenen Chargen oder Platten weisen häufig technische Variationen auf. Die Normalisierung ist ein entscheidender Schritt, der die Daten anpasst, um Messungen über Experimente hinweg vergleichbar zu machen, indem technisches Rauschen entfernt und gleichzeitig das biologische Signal erhalten bleibt.
Schritt 4: Merkmalsextraktion
Rohdaten liegen oft nicht in einem Merkmalsformat vor. Ein Bild muss beispielsweise verarbeitet werden, um numerische Merkmale wie Zellgröße, Form und Intensität zu extrahieren. Eine DNA-Sequenz könnte in einen k-mer-Frequenzvektor umgewandelt werden. Dieser Schritt wandelt komplexe Daten in Zahlen um, die die KI verwenden kann.
Schritt 5: Endgültige Zusammenstellung und Speicherung
Schließlich fügt die Pipeline die normalisierten Merkmale mit den standardisierten Metadaten zusammen. Dadurch wird die endgültige, bereinigte Merkmalsmatrix erstellt, die dann in einem stabilen, abfragbaren Format (wie Parquet oder einer Datenbank) zur Modellschulung gespeichert wird.
Die Abwägungen verstehen
Die Strukturierung von Daten ist kein neutraler Prozess. Jede Entscheidung, die Sie treffen, kann die Leistung und Interpretation des endgültigen Modells beeinflussen.
Überverarbeitung vs. Unterverarbeitung
Aggressive Normalisierung oder Filterung kann manchmal subtile, aber wichtige biologische Signale entfernen. Umgekehrt führt das Versäumnis, technisches Rauschen zu entfernen, dazu, dass Ihr Modell aus experimentellen Artefakten statt aus Biologie lernt. Dies ist eine ständige Balance.
Standardisierung erzeugt anfänglichen Mehraufwand
Die Implementierung von Data Governance erfordert erhebliche Anfangsanstrengungen und die Zustimmung des gesamten Teams. Es kann sich anfangs anfühlen, als würde es die Forschung verlangsamen, aber es zahlt sich massiv aus, indem es Monate der Aufräumarbeiten später verhindert.
Die Gefahr der Datenlecks (Data Leakage)
Eine entscheidende Funktion der Pipeline ist die getrennte Aufbewahrung von Trainings- und Testdaten. Wenn Informationen aus dem Testsatz (z. B. seine Gesamtverteilung) zur Normalisierung des Trainingssatzes verwendet werden, wird die Leistung Ihres Modells künstlich aufgebläht und es wird in der realen Welt versagen.
Die richtige Wahl für Ihr Ziel treffen
Ihr Ansatz zur Datenstrukturierung sollte von Ihrem ultimativen Ziel geleitet werden.
- Wenn Ihr Hauptaugenmerk auf Reproduzierbarkeit liegt: Priorisieren Sie von Anfang an eine strenge Data Governance und versionskontrollierte, vollständig automatisierte Pipelines.
- Wenn Ihr Hauptaugenmerk auf schnellem Prototyping liegt: Beginnen Sie mit einem kleinen, manuell kuratierten Datensatz, um Ihren KI-Ansatz zu validieren, bevor Sie in eine vollständige Pipeline investieren.
- Wenn Ihr Hauptaugenmerk auf der Skalierung in einer großen Organisation liegt: Investieren Sie stark in zentralisierte Datenspeicherung, gemeinsame Ontologien und gemeinsame Pipeline-Komponenten, um Datensilos zu verhindern.
Letztendlich ist die Behandlung Ihrer Daten mit der gleichen Sorgfalt wie Ihre Nasslaborexperimente die Grundlage für den Aufbau einer erfolgreichen und zuverlässigen biologischen KI.
Zusammenfassungstabelle:
| Schritt | Schlüsselaktion | Zweck |
|---|---|---|
| Data Governance | Festlegen von Namenskonventionen, Ontologien, Metadatenstandards | Sicherstellung von Konsistenz und Vergleichbarkeit über Experimente hinweg |
| Datenpipeline | Erfassen, Parsen, QC, Normalisieren, Merkmale extrahieren, Zusammenstellen | Automatisierung der Transformation von Rohdaten in eine KI-taugliche Merkmalsmatrix |
| Abwägungen | Ausgleich von Überverarbeitung vs. Unterverarbeitung, Verwaltung des Mehraufwands | Optimierung der Modellleistung und Vermeidung von Datenlecks |
Haben Sie Schwierigkeiten bei der Standardisierung Ihrer Nasslabor-Daten für KI? KINTEK ist spezialisiert auf Laborpressen, einschließlich automatischer Laborpressen, isostatischer Pressen und beheizter Laborpressen, die Labore dabei unterstützen, die Datenzuverlässigkeit und experimentelle Effizienz zu verbessern. Lassen Sie uns Ihnen helfen, konsistente Ergebnisse zu erzielen – kontaktieren Sie uns noch heute, um Ihre Anforderungen zu besprechen und herauszufinden, wie unsere Lösungen Ihre KI-gestützte Forschung unterstützen können!
Visuelle Anleitung
Ähnliche Produkte
- Manuell beheizte hydraulische Laborpresse mit integrierten Heizplatten Hydraulische Pressmaschine
- Hydraulische Laborpresse 2T Labor-Pelletpresse für KBR FTIR
- Automatische beheizte hydraulische Pressmaschine mit heißen Platten für das Labor
- Manuelle beheizte hydraulische Laborpresse mit heißen Platten
- Automatische beheizte hydraulische Hochtemperatur-Pressmaschine mit beheizten Platten für das Labor
Andere fragen auch
- Was sind die wichtigsten technischen Anforderungen an eine Heißpresse? Beherrschung von Druck und thermischer Präzision
- Was sind die Vorteile von Heißpressgeräten für Sulfidbatterieplatten? Erhöhen Sie die Dichte und reduzieren Sie die Impedanz
- Wie hilft eine hydraulische Presse beim Umformen von Materialien? Schalten Sie Präzision und Leistung für Ihre Laboranforderungen frei
- Welche Kernbedingungen bietet eine Laborhydraulikpresse? Optimierung der Heißpressung für 3-Schicht-Spanplatten
- Warum ist eine hochpräzise Temperaturregelung von Heizplatten in Laborhydraulikpressen für die Holzverdichtung entscheidend?



















